Analyse de variance à mesures répétées (repeated measures ANOVA)
Introduction
L'analyse de variance (ANOVA) à mesures répétées est un test paramétrique permettant de déterminer s'il existe une différence significative entre plusieurs mesures d'un même échantillon dans le temps.
Conditions d'application
Conditions d'application
Pour pouvoir être appliqué, ce test nécessite que les échantillons soient issus de distributions normales et que la sphéricité des variances soit respectée (variances similaires entre les différents points de mesure).
Pour vérifier si les distributions sont normales, nous devons utiliser un test de Shapiro-Wilk:
# Soit y la variable dépendante et time le facteur temporel (intra-sujet)
by(data$y, data$time, shapiro.test)
Information
Nous n'avons pas besoin de vérifier la sphéricité des variances, car le test est effectué directement dans la fonction de l'ANOVA, et la correction de Greenhouse-Geisser sera appliquée en cas de problème.
Exécution du test
# Soit y la variable dépendante,
# ID l'identifiant de chaque participant.e.x
# et time le facteur temporel (intra-sujet)
library(rstatix)
m <- anova_test(
data=data,
dv=y,
wid=ID,
within=time
)
get_anova_table(m, correction="GG")
Nous devons ensuite réaliser un test post-hoc, pour connaître entre quelles mesures cette différence s'exprime. Ici, nous utilisons des tests t sur mesures répétées:
pairwise.t.test(data$y, data$time, paired=TRUE, p.adjust.method="bonf")
Taille d'effet
Information
La taille d'effet est automatiquement calculée par la fonction que nous utilisons pour calculer l'ANOVA. Nous n'avons donc pas besoin de la calculer.
Exemple
Voici nos données et notre fichier d'analyse:
| Fichiers |
|---|
| repeated-measures-anova.csv |
| repeated-measures-anova.R |
Commençons par activer les librairies nécessaires dans R:
library(rstatix)
library(RcmdrMisc)
Puis, importons nos données:
data <- read.csv("repeated-measures-anova.csv")
df <- data.frame(
ID=rep(data$ID, 3),
score=c(data$T1, data$T2, data$T3),
time=factor(rep(c("T1", "T2", "T3"), each=100))
)
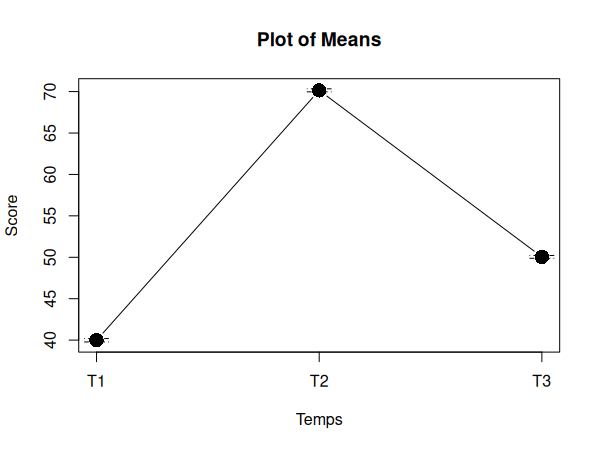
Nous pouvons maintenant visualiser nos données dans un diagramme:
plotMeans(df$score, df$time, xlab="Temps", ylab="Score")
Vérifions maintenant la normalité des distributions:
by(df$score, df$time, shapiro.test)
| W | p | |
|---|---|---|
| T1 | 0.98399 | 0.2678 |
| T2 | 0.98429 | 0.2822 |
| T3 | 0.98882 | 0.5705 |
Puisque les conditions d'applications sont satisfaites, nous pouvons réaliser une ANOVA à mesures répétées:
m <- anova_test(
data=df,
dv=score,
wid=ID,
within=time
)
get_anova_table(m, correction="GG")
| Effect | Dfn | Dfd | F | p | ges |
|---|---|---|---|---|---|
| time | 1.99 | 197 | 6591.602 | 5.66e-181 | 0.979 |
Nous pouvons conclure qu'il y a un effet, et que cet effet est grand. Il ne nous reste maintenant plus qu'à chercher entre quelles mesures s'exprime cet effet:
pairwise.t.test(df$score, df$time, paired=TRUE, p.adjust.method="bonf")
| T1 | T2 | |
|---|---|---|
| T2 | <2e-16 | - |
| T3 | <2e-16 | <2e-16 |
Il existe donc une différence significative entre toutes les mesures. Nous pouvons donc conclure qu'il existe une grande différence significative entre nos mesures (F~(1.99, 297)~ = 6591.6, p<.001, η2G = 0.98).